

今天为大家介绍的是来自Hailong Li,Ka-Chun Wong和Hengtong Zhang团队的一篇论文。基于结构的生成方法在计算机辅助药物发现中至关重要,它通过探索广阔的化学空间来设计与靶点高亲和力结合的配体。然而,传统的计算机模拟方法受限于计算效率低下,而机器学习方法则因自回归采样面临瓶颈。为了解决这些问题,作者开发了一种条件深度生成模型PMDM,用于生成符合特定靶标的三维分子。

最近,扩散模型在计算机视觉任务中受到了极大的关注,特别是在点云生成方面,这与三维分子生成有诸多相似之处。这些方法通过学习联合分布,擅长于三维对象的内部填充。尽管已经开发了一种用于基于结构的分子生成的扩散模型,但它需要训练用户定义的参数,导致采样效率低下。此外,它仅使用完全连接的邻接矩阵,因而忽略了分子图的内在拓扑结构。受计算机视觉任务中扩散模型成功的启发,作者提出了一个名为“口袋基分子扩散模型”(PMDM)的一站式生成框架来解决这些问题。图1概述了PMDM的概览。具体来说,固定口袋信息的分子原子被视为三维点云,并在前向过程中扩散,这与非平衡热力学中的现象类似。PMDM的目标是学习如何逆转这一过程来模拟条件数据分布。然而,如果将三维分子几何结构表达为三维点云,常规方法无法涉及边信息,如化学键信息。因此作者定义了一种双扩散策略来构建两种类型的虚拟边。具体来说,原子对的原子间距离低于一定阈值时,通过共价定位边缘连接,因为当两个原子足够接近时,化学键可以主导原子间力,而全局边连接到其余原子对,以模拟范德华力。此外,作者设计了一个服从分子几何系统的平移、旋转、反射和排列等变的动态核。在合成的CrossDocked数据集上的实验表明,PMDM能够生成具有高结合亲和力的药物样、易合成、多样化的分子,并在多个评估指标上超越最先进的模型。
模型架构
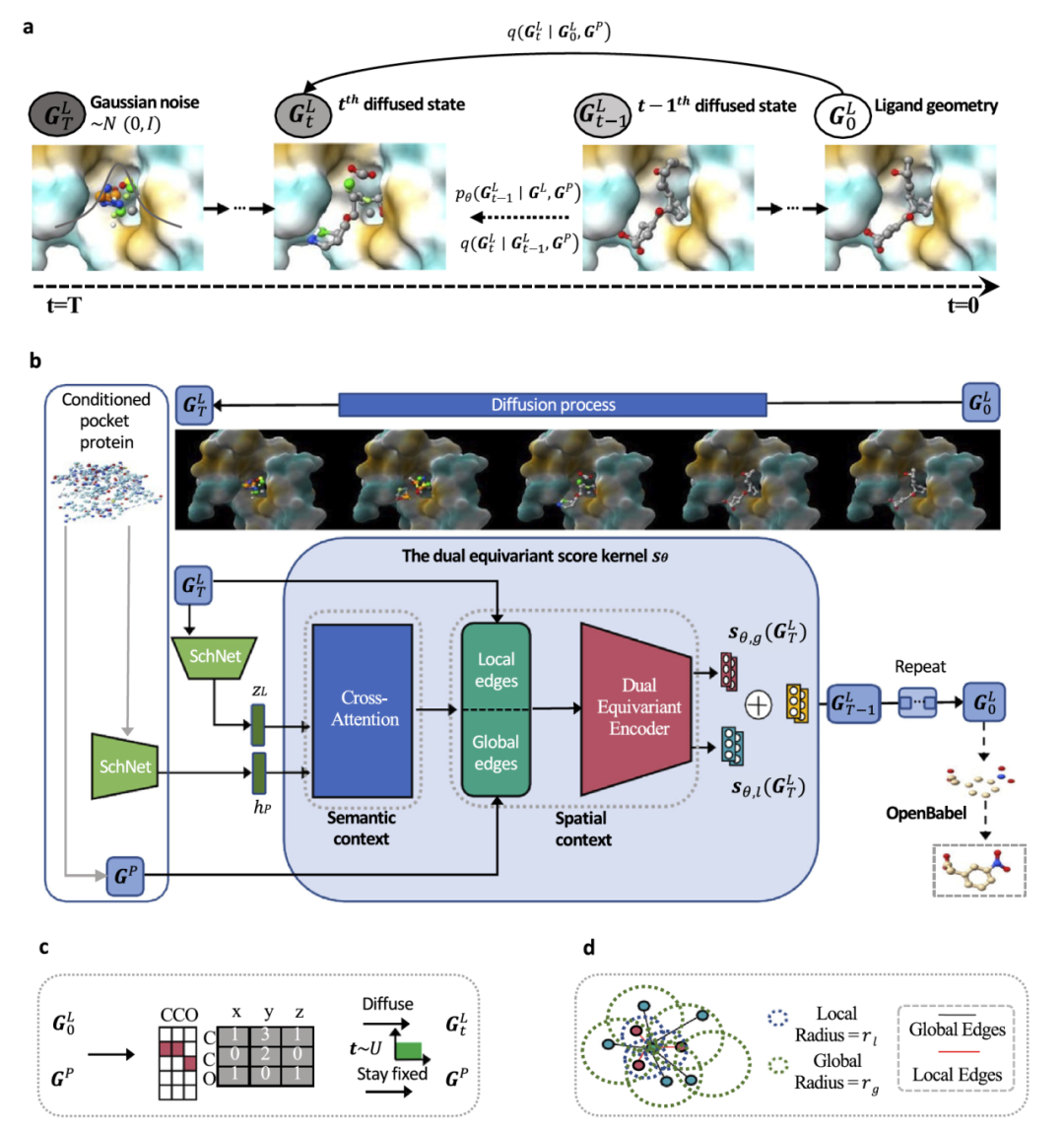
图 1
图1概述了条件生成模型PMDM的整体架构,阐明了其结构组件以及在训练和采样过程中涉及的各个步骤。PMDM在前向过程中逐渐引入高斯噪声,同时利用参数化的逆过程来迭代地消除噪声(图1a)。该模型包括两个不变的图神经网络Schnet,用于获取分子嵌入zL和口袋嵌入hP(图1b)。为了促进条件生成,作者设计了两种上下文机制来结合蛋白口袋的语义和几何信息。PMDM使用交叉注意力层来计算分子和蛋白、蛋白口袋的注意力得分。此外,采用了双扩散策略来使模型能够识别原子级力。这种策略涉及构建两种类型的虚拟边。首先,原子间距离低于局部阈值τl的原子对通过共价局部边缘连接,因为当原子靠近时,化学键倾向于主导原子间力。其次构建了全局边,这些边连接到其余的原子对,用以模拟原子间距大于局部阈值τl但小于全局阈值τg的范德华力(图1d)。此外设计了一个等变动态核,遵循分子几何系统的平移、旋转、反射和排列等变性。为了确保生成的分子适应结构口袋,在双等变编码器中更新隐藏状态时保持口袋位置固定。
通用评价指标上评估PMDM
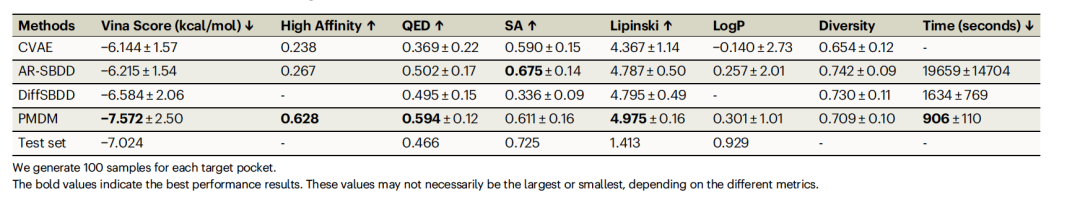
表 1
作者为测试集中的每个目标蛋白生成了100个分子(总计10000个分子)。这里,生成分子的大小是从训练集的大小分布中采样得到的。PMDM和基线模型的整体结果展示在表1中。除了SA和多样性之外,PMDM在几乎所有指标上都优于所有基线模型。根据Vina分数,PMDM能够生成与口袋高亲和力的分子(-7.472 ± 2.90),比最好的自回归基线模型AR-SBDD好20.2%,比另一个扩散模型DiffSBDD好15.0%。此外,PMDM在QED(0.594 ± 0.12)上比AR-SBDD和DiffSBDD分别高出18.3%和20.0%,在Lipinski(4.975 ± 0.16)上分别高出3.9%和3.7%。PMDM的logP值在合规范围内(-0.4 ~ 5.6),表明PMDM生成的分子更有作为药物候选物的潜力,这对临床试验至关重要。
对PMDM在局部几何结构上的分析

图 2
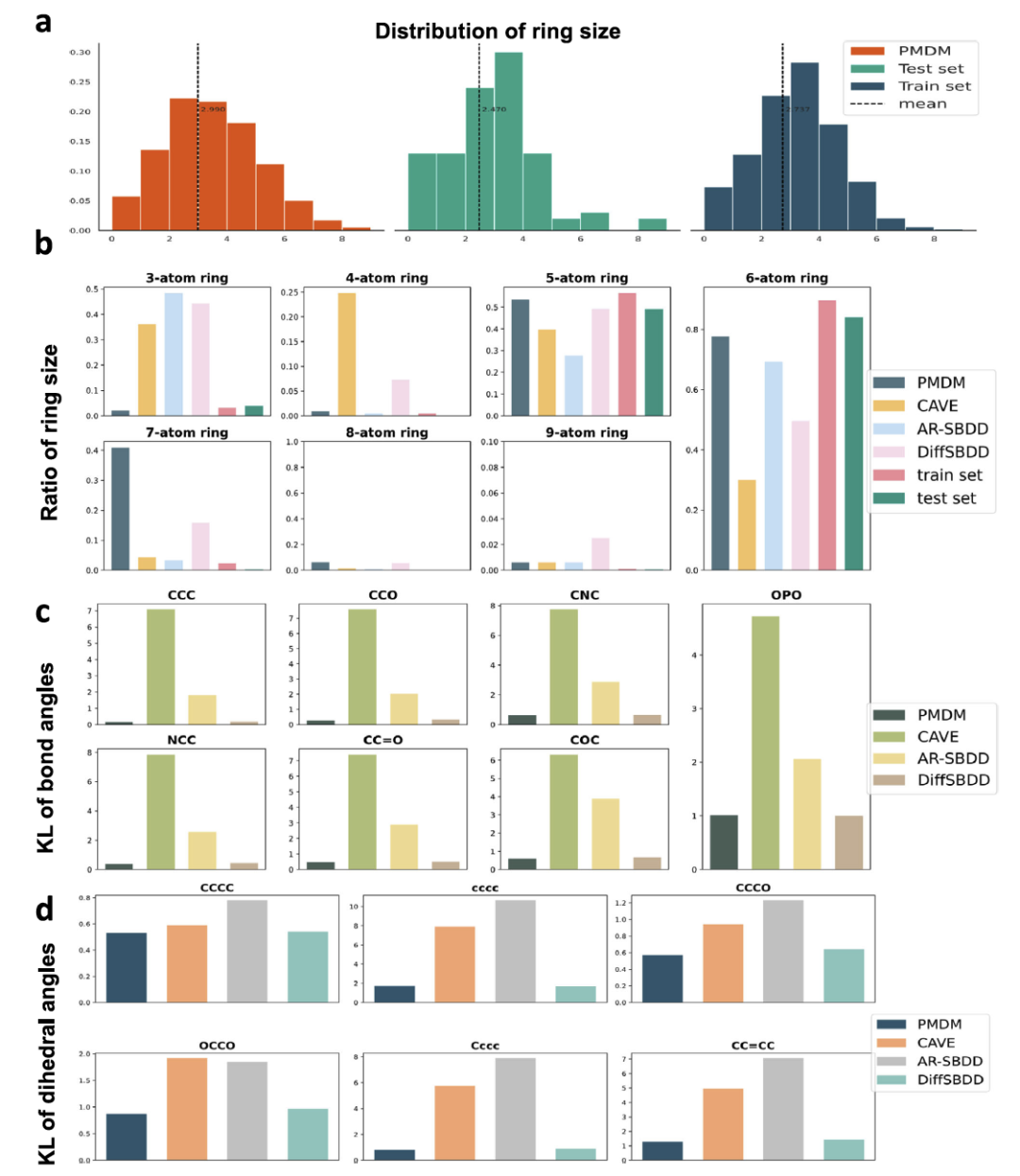
图 3
虽然传统指标在一定程度上可以反映生成分子的质量,但在评估模型性能时,还需要考虑生成分子的子结构的质量。作者选择几种口袋蛋白作为子结构分析的代表性样本进行可视化。如图2所示。可以观察到,AR-SBDD和DiffSBDD倾向于生成三原子环,而模型PMDM避免生成这种不稳定的环。尽管数据集中只含有3%的三原子环,AR方法生成了更多这种不稳定的结构,这意味着这些方法陷入了局部最优,并未能很好地学习数据分布。相反,PMDM能够考虑口袋孔的形状,生成更大且更复杂的环,这在3AF2口袋样本中有所展示。为了全面了解生成分子的结构分布,作者展示了PMDM生成的分子与测试集和训练集中分子的环数分布(图3a)。PMDM的分布接近于测试集和训练集。PMDM生成的分子平均含有2.990个环,而测试集和训练集中的分子平均含有2.470个环和2.737个环。总体来说,结果表明PMDM能够从局部角度学习环子结构的尺寸分布,并从全球角度学习环数的分布。为了进一步量化这些方法生成的分子中环子结构的比例,作者报告了训练集、测试集以及这些方法生成的集合中含有不同大小环的分子比例。在含有多个环的分子的情况下,计数过程会考虑每一个存在的独立环,从而导致重复计数与环数成比例。如图3b所示,PMDM生成的分子中不稳定环较少,包括三原子环和四原子环。自回归方法倾向于仅考虑先前生成的部分,限制自身于局部拓扑结构,这通常导致生成小环。DiffSBDD为所有原子构建完全连接的边,可能由于原子间距减小,导致形成小环的可能性更高。
分析PMDM在化学空间分布上的表现
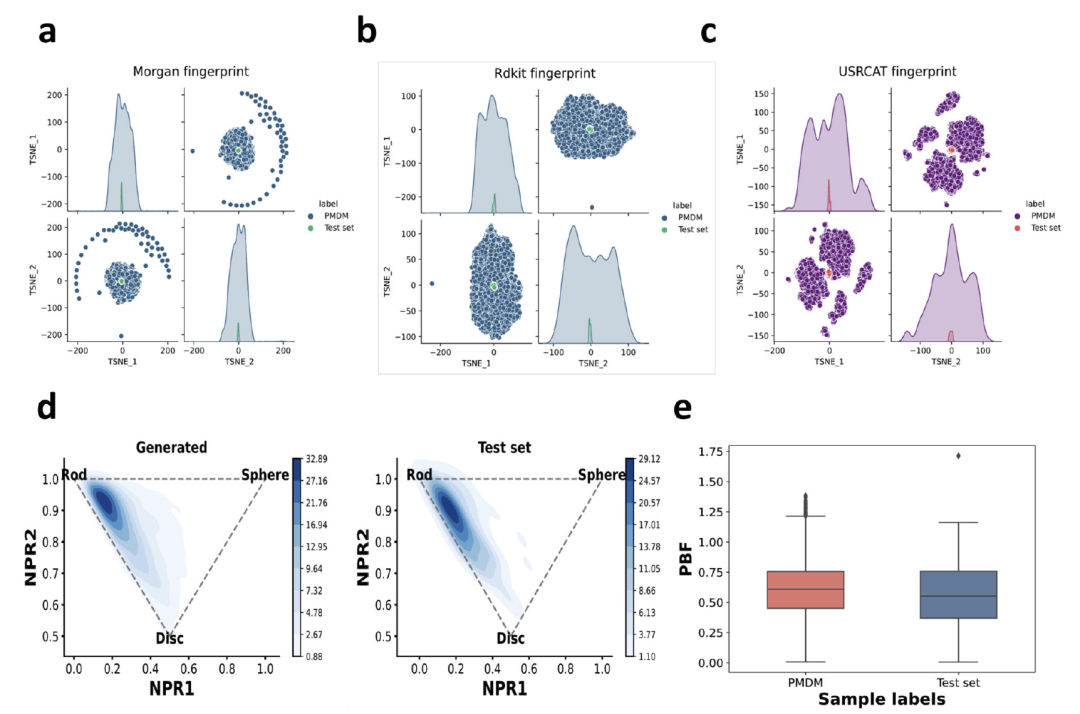
图 4
在分析了PMDM生成的分子的局部几何结构后,作者接着从全局角度评估生成的分子化学空间分布。由于化学结构的三维性是药物化学分子设计的本质,作者也关注化学结构的形状。在此采用包括Morgan、RDKit和USRCAT指纹的2D和3D分子指纹来表示生成分子和测试集分子的化学空间。使用t-SNE可视化化学空间分布如图4所示。PMDM生成的分子的化学空间可以覆盖测试集中的分子在2D亚结构空间中,表明PMDM能够正确地模拟测试集的2D化学空间(图4a, b)。如图4c所示,生成分子的3D化学空间基本上可以捕捉到测试分子的空间,这归功于构象的复杂性。尽管未能完全覆盖参考化学空间,生成分子和测试集分子之间没有显著的分布不匹配。
引导生成和优化
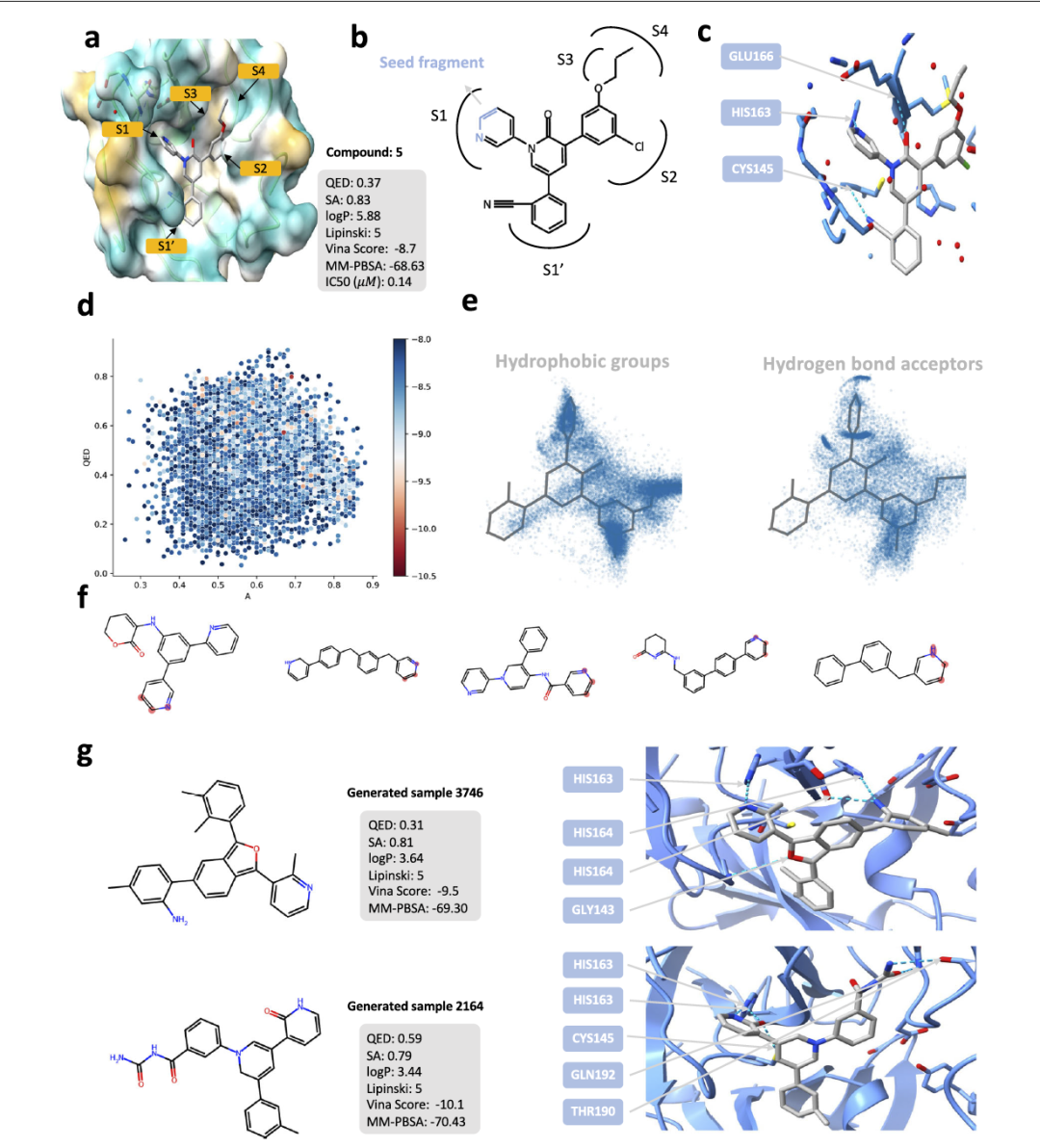
图 5
PMDM使得针对特定靶标的生物活性分子生成成为可能。为了进一步探讨PMDM的实际应用,作者使用训练好的模型来生成针对SARS-CoV-2相关蛋白具有高亲和力的分子。在这里选择SARS-CoV-2的主要蛋白酶(Mpro)作为测试案例,按照先前的工作进行非共价抑制剂的设计。SARS-CoV-2中的Mpro是主要的蛋白酶,可以在多个位置切割多聚蛋白,使其成为一个可行的药物靶标。作者的目标是生成具有更多样化框架的分子,这称为引导生成。为此,作者使用三个原子作为种子片段,即图5b的蓝色部分。采用填充方法根据时间步骤扩散种子片段的数据,并与生成部分组合。生成了40000个分子,并筛选出Vina分数低于-8.0 kcal/mol的分子。最终获得了10627个具有高亲和力的分子。检查了所有过滤后的分子,没有一个出现在训练集中。这表明尽管参考分子的亲和力很高,PMDM仍能生成与目标蛋白良好结合的分子。如图5所示,作者绘制了过滤分子的三个关键属性(QED、SA和Vina分数)的分布。可以观察到,PMDM能够在保持良好属性的同时生成具有良好亲和力的分子。从统计上看,分子的平均QED值为0.57,高于参考化合物5,最高QED值为0.75。对于Vina分数,平均值为-8.6,最小值为-12.3,尽管在合成可及性方面表现欠佳,平均SA值为0.30,最大SA值为1.0。结果表明PMDM能够学习训练数据的分布。因此,它可以生成适应口袋结构且满足高药物样性和良好合成可及性要求的分子,无需将所需属性作为条件信息输入。

图 6
PMDM 使得在真实的合成生物活性分子设计中进行支架跳跃和连接器生成成为可能。在引导优化中,适当的活性化合物的支架跳跃非常重要。为了验证模型是否可以应用于支架跳跃以提高给定基本生物活性分子的结合亲和力,作者选择CDK2作为目标蛋白来生成具有核心结构的期望分子。作者使用PMDM对与CDK2复合的化合物13(PDB ID: 8H6T)进行支架跳跃,以开发潜在的抑制剂。参考化合物13在图6a中展示。化合物的吡啶部分朝向溶剂可接触区域,并未与CDK2表现出任何显著的极性或非极性接触。经过化学专家审核后,作者移除了吡啶环(图6a中的虚线框)并保留了剩余的片段作为种子支架(图6a),这是现有CDK2抑制剂的关键支架。最终利用PMDM生成了10000个分子以替换关键片段。然后通过Vina对接和MM-PBSA值进行潜在抑制剂的筛选,并进行视觉选择。最后选择了四种化合物进行进一步的视觉检查、合成和测试。如图6b所示,所有潜在抑制剂的Vina分数和MM-PBSA值较高,SA分数适中。进行体外实验以测试它们的CDK1/2抑制活性。如图6b所报告的,所有分子在酶测定中显示出改善的CDK2活性,CDK1选择性至少达到~44倍。化合物6793在吡啶上重新引入了一个氰基,表现出最佳的CDK1选择性(124倍)。值得注意的是,含有羟甲基的吡嗪环的化合物6849表现出最高的CDK2活性和相当的CDK1选择性。此外,化合物6849在引导优化活动中被证明是一个很好的先导分子,并表现出对其他密切相关的激酶的良好选择性,包括CDK9(CDK9/T1抑制IC50 = 32.3 nM,CDK9/CDK2 = 127)和GSK3β(GSK3β抑制IC50 = 703 nM,GSK3β/CDK2 = 2780)。
编译 | 曾全晨
审稿 | 王建民
参考资料
Huang, L., Xu, T., Yu, Y., Zhao, P., Chen, X., Han, J., ... & Zhang, H. (2024). A dual diffusion model enables 3D molecule generation and lead optimization based on target pockets. Nature Communications, 15(1), 2657.